1/ Profilage pharmacologique computationnel (A-C. Camproux & O. Taboureau, resp, A. Badel, D. Flatters, C. Geneix, M. Petitjean, L. Regad) :
Mots Clés: polypharmacologie, Interaction protéine-petites molécules, biostatistique, bioinformatique, intégration de données, pharmacologie et toxicologie des systèmes, réseaux biologique.
Les recherches principales de ce thème à l’aide de l’utilisation et du développement d’outils biostatistique, bioinformatique et des réseaux biologiques concernent :
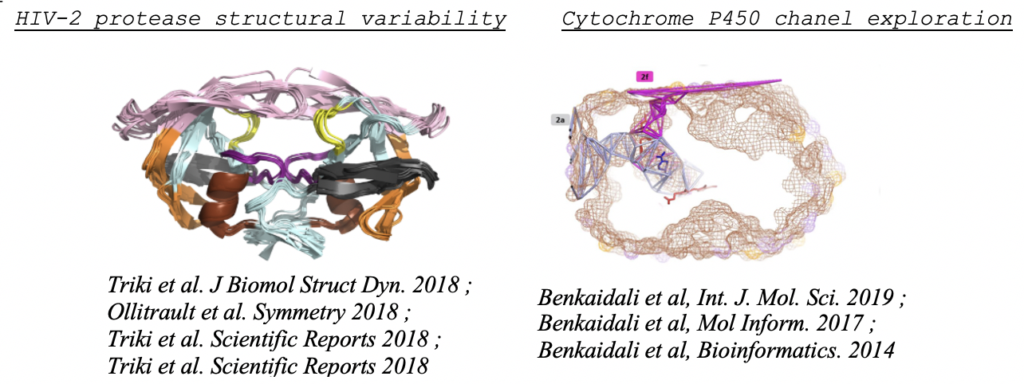
 – Analyse structurale des cibles protéiques et modélisation des interactions cible-ligand: Nous poursuivons l’analyse structurale des structures protéiques en prenant en compte leur flexibilité afin de prioriser les cibles thérapeutiques médicamenteuses prometteuses ; et la prédiction statistique des interactions médicament-cible. Nous développons des protocoles pour explorer les cibles thérapeutiques en (i) étudiant les impacts de mutations ponctuelles sur la structure (site de liaison et région d’interface), à l’aide d’approches de dynamique moléculaire et de bioinformatique structurale et (ii) en développant des méthodes biostatistiques pour la prédiction des partenaires des interactions médicament-cible et de leurs profils. Nous nous concentrons notamment sur la prédiction in silico de petites molécules pour une cible donnée et les effets indésirables et hors cibles. Nos protocoles seront appliqués à différents systèmes, tels que la protéase du VIH-2, à la protéine non structurales (NS1/NS2) du virus de la grippe, à des enzymes et des cibles de toxicologie en collaboration avec des expérimentateurs et experts.
– Analyse structurale des cibles protéiques et modélisation des interactions cible-ligand: Nous poursuivons l’analyse structurale des structures protéiques en prenant en compte leur flexibilité afin de prioriser les cibles thérapeutiques médicamenteuses prometteuses ; et la prédiction statistique des interactions médicament-cible. Nous développons des protocoles pour explorer les cibles thérapeutiques en (i) étudiant les impacts de mutations ponctuelles sur la structure (site de liaison et région d’interface), à l’aide d’approches de dynamique moléculaire et de bioinformatique structurale et (ii) en développant des méthodes biostatistiques pour la prédiction des partenaires des interactions médicament-cible et de leurs profils. Nous nous concentrons notamment sur la prédiction in silico de petites molécules pour une cible donnée et les effets indésirables et hors cibles. Nos protocoles seront appliqués à différents systèmes, tels que la protéase du VIH-2, à la protéine non structurales (NS1/NS2) du virus de la grippe, à des enzymes et des cibles de toxicologie en collaboration avec des expérimentateurs et experts.
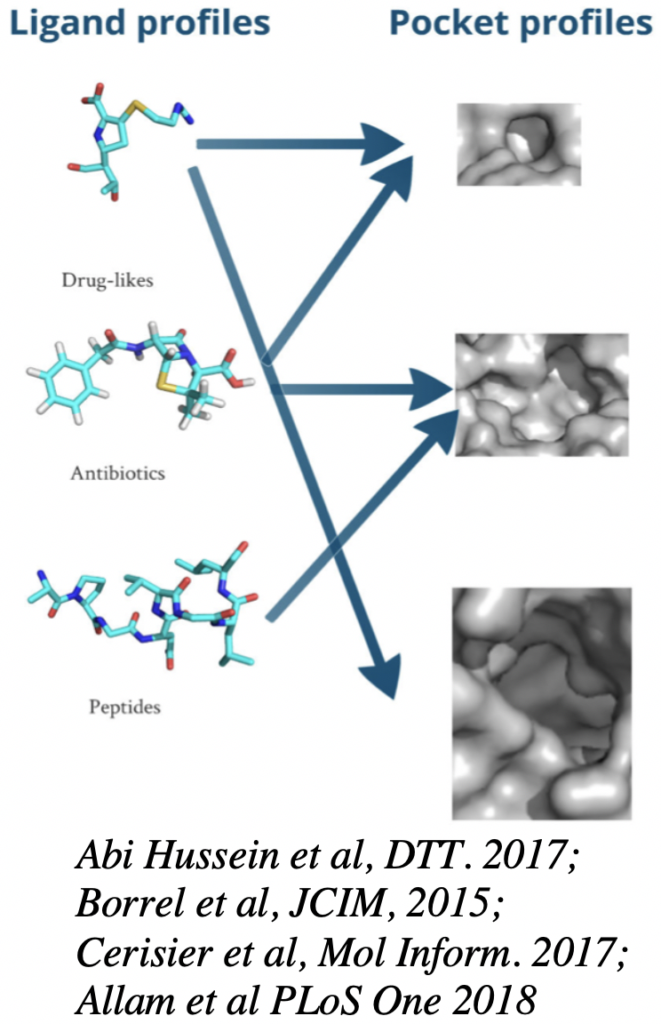
– La modélisation des interactions protéines-ligands permettant la découverte de hits thérapeutique, de déterminer les mécanismes de liaisons entre une petite molécule et une (ou plusieurs) cible(s) protéique et donc d’estimer son profil pharmacologique. Nous nous intéresserons notamment à des enzymes du métabolisme et potentiellement intéressante pour traiter la trisomie 21 (CBS, Dirk1A).
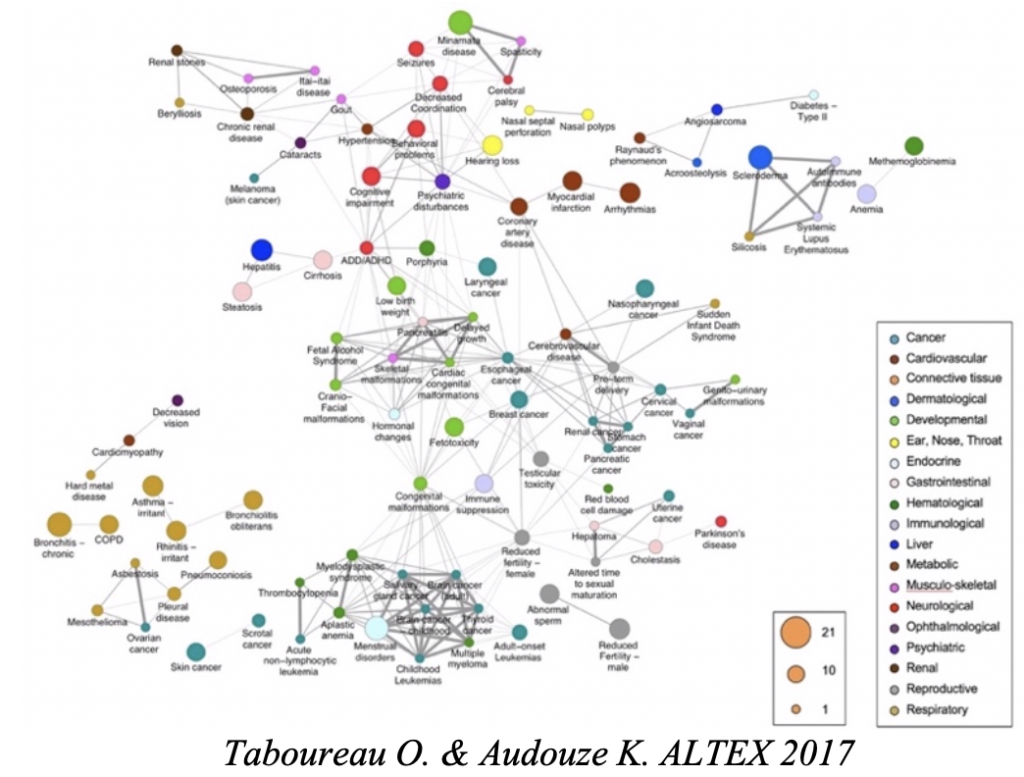
– L’analyse des relations ligand-phenotype. L’objectif est d’analyser l’impact des petites molécules à différents niveaux de complexité, aussi bien moléculaire, cellulaire, tissulaire qui peuvent expliquer l’apparition d’un phénotype donnée (maladie, effet secondaires…). Nous développons et exploitons des méthodes de réseaux biologiques ainsi que biostatistique qui s’avèrent pertinentes dans l’identification de mécanisme impliqués dans certaines pathologies du foie (steatose, lésion hépatique), du diabète ou de la reproduction.
2/ Interactions Peptide-protéine et design de peptides sondes ou candidats (P. Tufféry, resp., G. Moroy, S. Murail, J. Rey, S. De Vries) :
Mots Clés: Interactions protéine-peptides, conception de peptides à vocation thérapeutique, méthodologie, structure-based peptide design, peptides interférents.
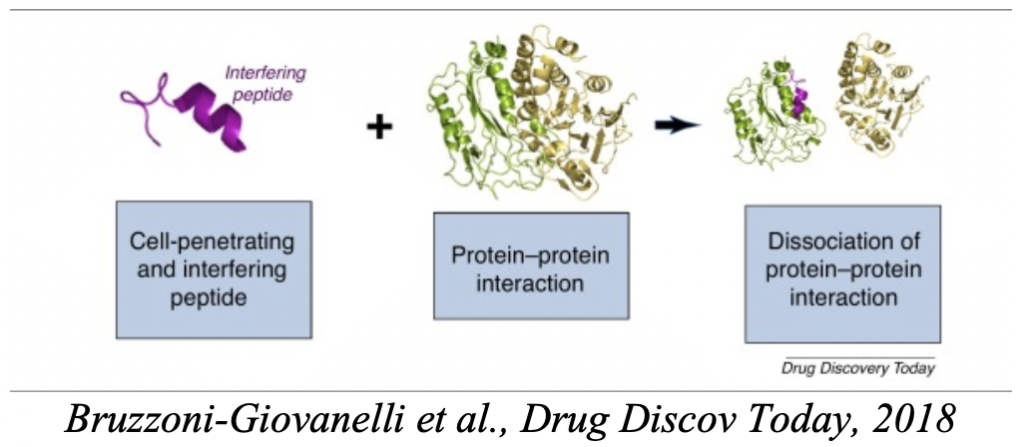
Notre domaine d’application principal – mais non exclusif – est la conception de peptides interférants d’interactions protéine-protéine cibles dans un contexte de régulation du métabolisme, ou de lutte contre le dérèglement cancéreux. Cette activité est soutenue par des développements méthodologiques sur les directions suivantes:
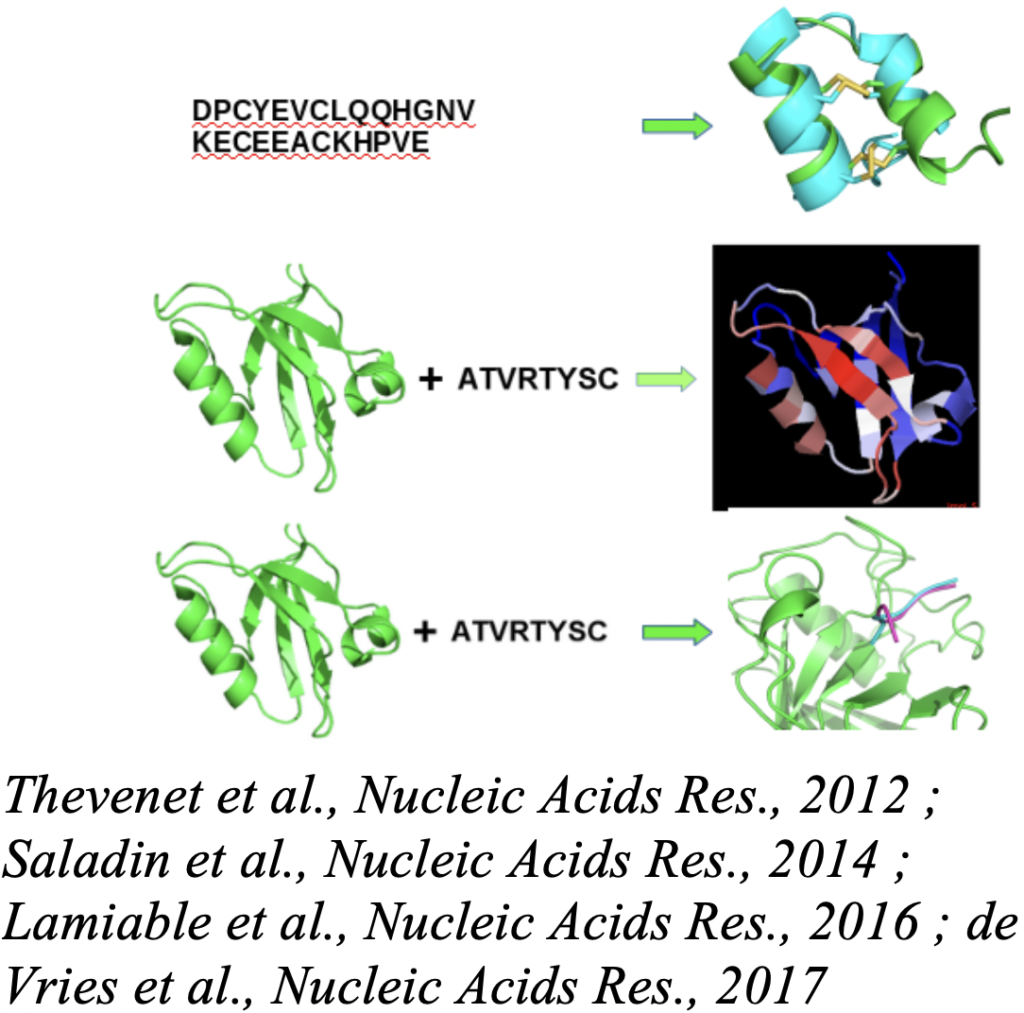 – Développement de peptides sondes ou à vocation thérapeutique implique le développement de protocoles in silico spécifiques. Nous nous intéressons à l’étude des interactions peptide-protéine guidée par la structure de la protéine (PEP-FOLD, PEP-SiteFinder, pepATTRACT), avec un focus sur l’amarrage peptide-protéine (hots spots, échantillonnage conformationnel, scoring), sur des approches d’optimisation des peptides (cyclisation – PEP-Cylizer, variants de séquence). Nous nous intéressons également au couplage d’approches in silico à la technique in vitro PEPscan pour l’identification de peptides interférents des interactions protéines-protéines.
– Développement de peptides sondes ou à vocation thérapeutique implique le développement de protocoles in silico spécifiques. Nous nous intéressons à l’étude des interactions peptide-protéine guidée par la structure de la protéine (PEP-FOLD, PEP-SiteFinder, pepATTRACT), avec un focus sur l’amarrage peptide-protéine (hots spots, échantillonnage conformationnel, scoring), sur des approches d’optimisation des peptides (cyclisation – PEP-Cylizer, variants de séquence). Nous nous intéressons également au couplage d’approches in silico à la technique in vitro PEPscan pour l’identification de peptides interférents des interactions protéines-protéines.
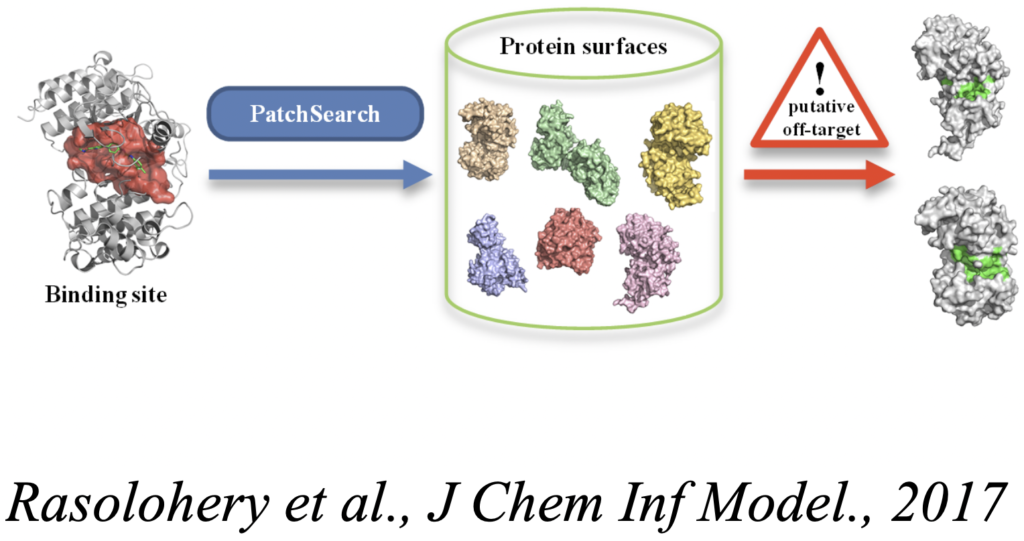 – Inférence de connaissance par fouille de données structurales. L’analyse à grande échelle des structures protéiques disponibles constitue un réservoir de données à haute valeur ajoutée pour l’étude de la sélectivité des interactions et le développement d’approches de peptides spécifiques d’interactions cibles. Nous nous intéressons aux similitudes de conformations de fragments protéiques ou de patches de surface (PatchSearch), et leurs applications à la conception de peptides sélectifs de cibles d’intérêt.
– Inférence de connaissance par fouille de données structurales. L’analyse à grande échelle des structures protéiques disponibles constitue un réservoir de données à haute valeur ajoutée pour l’étude de la sélectivité des interactions et le développement d’approches de peptides spécifiques d’interactions cibles. Nous nous intéressons aux similitudes de conformations de fragments protéiques ou de patches de surface (PatchSearch), et leurs applications à la conception de peptides sélectifs de cibles d’intérêt.
3/ Plate-forme RPBS (« Ressource Parisienne en Bioinformatique Structurale »)
 L’équipe héberge la plate-forme RPBS (bioserv.rpbs.univ-paris-diderot.fr) qui propose service et prestation en bioinformatique structurale (analyse et modélisation de la structure de protéines et des complexes protéine-protéine) et développement de composés à vocation thérapeutique (docking/criblage de petits composés à vocation thérapeutique, docking/optimisation de peptides). Cette plate-forme IBiSA est membre de l’Institut Français de Bioinformatique (www.france-bioinformatique.fr) et de ChemBioFrance (www.chembiofrance.fr).
L’équipe héberge la plate-forme RPBS (bioserv.rpbs.univ-paris-diderot.fr) qui propose service et prestation en bioinformatique structurale (analyse et modélisation de la structure de protéines et des complexes protéine-protéine) et développement de composés à vocation thérapeutique (docking/criblage de petits composés à vocation thérapeutique, docking/optimisation de peptides). Cette plate-forme IBiSA est membre de l’Institut Français de Bioinformatique (www.france-bioinformatique.fr) et de ChemBioFrance (www.chembiofrance.fr).